Dplyr

El paquete dplyr ofrece un conjunto de herramientas para la manipulación de datos, aunque estas tareas también pueden ser llevadas a cabo con el paquete base de R, la simplicidad que provee dplyr facilita y agilizara tales operaciones.
Las funciones principales que provee el paquete dplyr son:
select: devuelve solo las columnas indicadas.filter: permite filtrar filas según una expresión lógica.arrange: ordena las filas en función de los valores de una o más columnas.rename: permite cambiar el nombre de una columna.mutate: permite agregar una nueva columna o transformar una existente.distinct: remueve las filas con valores duplicados.%>%: permite realizar resúmenes estadísticos de variables.summarise: permite realizar resúmenes estadísticos de variables.
Para mostrar el funcionamiento de dplyr descargaremos 1000 registros para el género Mammillaria de la base de datos GBIF (the Global Biodiversity Information Facility).
## Cargar el paquete rgbif
library(rgbif)
## Obtener 1000 registros de GBIF para el género Mammillaria
Mammillaria <- occ_search(scientificName = "Mammillaria", limit = 1000)
## Asignar a una variable el componente data de la lista Mammillaria
Mammillaria_gbif <- Mammillaria$data
select()
La base de datos de Mammillaria contiene 1000 observaciones y 92 variables, a veces no es necesario trabajar con todas las variables y podemos seleccionar solo aquellas con las que deseamos trabajar para simplificar nuestra base de datos.
## Cargar el paquete dplyr
library(dplyr)
## Seleccionar las variables con las que vamos a trabar y asignamos el nuevo conjunto a Mammillaria_1
Mammillaria_1 <- select(Mammillaria_gbif, family, species, decimalLatitude, decimalLongitude, country, stateProvince, year, month, day, verbatimLocality, recordedBy, identifiedBy)
filter()
Continuando con la depuración de nuestra base de datos, posiblemente tendremos que filtrar filas para conservar a las que satisfacen las condiciones dadas. Para conservarse, la fila debe producir un valor de VERDADERO para todas las condiciones. Existen operadores y funciones que son útiles al construir las expresiones utilizadas para filtrar los datos:
| Operador | Descripción |
|---|---|
< |
menor que |
<= |
Menor o igual |
> |
Mayor que |
>= |
Menor o igual |
== |
Igual |
!= |
Distindo de |
%in% |
Identificar si un elemento pertenece a un vector o data frame |
## Filtrar las especies de Mammillaria que esten en el estado de Puebla
Mammillaria_2 <- filter(Mammillaria_1, stateProvince == "Puebla")
## Filtrar las especies de Mammillaria que esten en latitudes menores a 21°
Mammillaria_3 <- filter(Mammillaria_1, decimalLatitude < 21)
## Filtrar las especies de Mammillaria haageana, Mammillaria carnea y Mammillaria haageana
## Crear un vector con las especies antes mencionadas
Mammillaria_vec <- c("Mammillaria karwinskiana", "Mammillaria carnea", "Mammillaria haageana")
## Utilizar %in% para filtrar las especies contenidas en el vector Mammillaria_vec
Mammillaria_4 <- filter(Mammillaria_1, species %in% Mammillaria_vec)
Muchas veces nuestras bases de datos tendrán datos ausentes (NAs) y será necesario eliminar esos registros.
## Eliminar NAs de decimalLatitude con ayuda de la funcion is.na()
Mammillaria_5 <- filter(Mammillaria_1, !is.na(decimalLatitude))
En el caso de que tengamos múltiples argumentos para hacer un filtrado podemos utilizar los operadores boléanos como lo muestra la siguiente imagen:
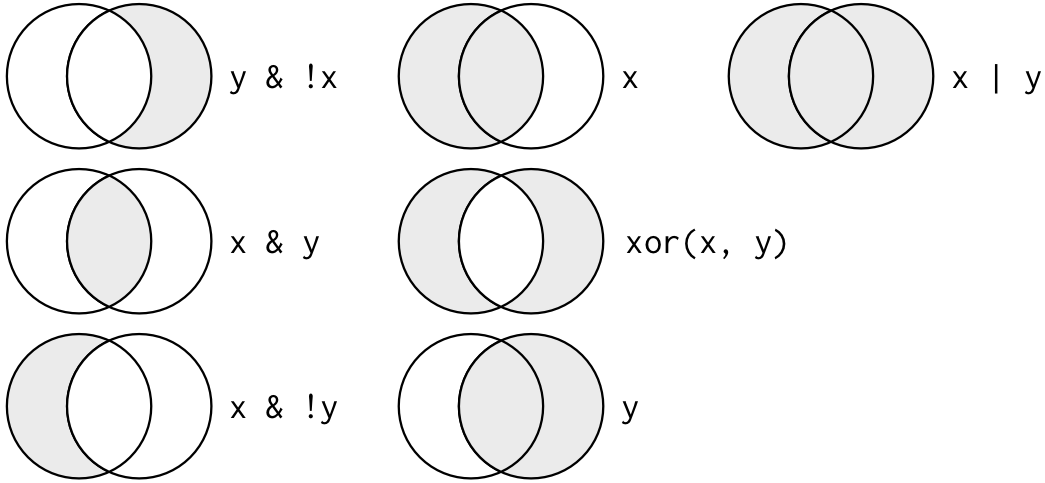
Por ejemplo, supongamos que x es la especie Mammillaria haageana y que y es el estado de Oaxaca.
y & !x: que el estado se igual a Oaxaca pero distinto de Mammillaria haageana.x: que la especie sea igual a Mammillaria haageana.x | y: que la especie sea igual a Mammillaria haageana o que el estado se igual a Oaxaca.x & y: que la especie sea igual a Mammillaria haageana y que el estado se igual a Oaxaca.xor(x, y): devolverá todas las filas donde solo se cumpla una de las condiciones, y no cuando se cumplan ambas condiciones.x & !y: que la especie sea igual a Mammillaria haageana pero distinto de Oaxaca.x: que el estado se igual a Oaxaca.
## Filtrar las filas que sean iguales al estado de Oaxaca y a la especie Mammillaria haageana
Mammillaria_6 <- filter(Mammillaria_1, stateProvince == "Oaxaca" & species == "Mammillaria haageana")
## Filtrar las filas que sean iguales al estado de Oaxaca o a la especie Mammillaria haageana
Mammillaria_7 <- filter(Mammillaria_1, stateProvince == "Oaxaca" | species == "Mammillaria haageana")
arrange()
También podemos ordenar las filas por los valores de las columnas seleccionadas.
## Ordenar de forma ascendente la latitud
Mammillaria_8 <- arrange(Mammillaria_1, decimalLatitude)
## Ordenar de forma descendente la latitud
Mammillaria_9 <- arrange(Mammillaria_1, desc(decimalLatitude))
## Ordenar de forma ascendente por especie y la latitud
Mammillaria_10 <- arrange(Mammillaria_1, species, decimalLatitude)
rename()
Al igual que en la base de R podemos cambia los nombres de variables o columnas.
## Cambiar el nombre de la variable family y species
Mammillaria_11 <- rename(Mammillaria_1, Familia = family, Especie = species)
mutate()
Se pueden agregar nuevas variables a la base de datos a partir de las ya existentes.
## Crear una funcion para convertir grados decimales a grados, minutos y segundos
dd2dms <- function(d1, var){
left1 <- d1 * 3600
degs <- trunc(left1/3600)
left2 <- left1 - degs * 3600
mins <- trunc(left2/60)
osec <- left2 - mins * (60)
if (var == "d"){
return(degs)
} else if (var == "m"){
return(mins)
} else {
return(osec)
}
}
## Convertir latitud en grados decimales a grados, minutos y segundos
Mammillaria_12 <- mutate(Mammillaria_1, Lat_gra = dd2dms(decimalLatitude, "d"),
Lat_min = dd2dms(decimalLatitude, "m"),
Lat_seg = dd2dms(decimalLatitude, "s"))
distinct()
Sirve para eliminar filas duplicadas y quedarnos con filas únicas.
## Eliminar los estados repetidos y quedarnos con solo un solo estado por fila
Mammillaria_13 <- distinct(Mammillaria_1, stateProvince)

El paquete magrittr ofrece un conjunto de operadores que hacen que su código sea más legible. En informática, una tubería (pipeline) consiste en una cadena de procesos conectados de forma tal que la salida de un proceso es la entrada del próximo. Dentro de magrittr el operador %>% es el utilizado como tubería para conectar varios procesos en uno.
## Utilizar el operador %>% para seleccionar columnas,
## filtrar, crear nuevas variable y eliminar filas dulicadas
Mammillaria_14 <- Mammillaria_gbif %>%
select(family, species, decimalLatitude, decimalLongitude,
country, stateProvince, year, month, day, verbatimLocality,
recordedBy, identifiedBy) %>%
filter(stateProvince == "Puebla" & species == "Mammillaria haageana") %>%
mutate(Lat_gra = dd2dms(decimalLatitude, "d"),
Lat_min = dd2dms(decimalLatitude, "m"),
Lat_seg = dd2dms(decimalLatitude, "s")) %>%
distinct(decimalLatitude, .keep_all = TRUE)
group_by(), count() y summarise()
group_by() toma un data frame o un tibble existente y la convierte en una conjunto agrupado donde las operaciones se realizan "por grupo". count() es una función que se utiliza para hacer conteos o sumas (n) utilizando a group_by() para realizar las operaciones. summarise() crea un nuevo marco de datos que tendrá una (o más) filas para cada combinación de variables de agrupación dado por group_by(); si no hay variables de agrupación, la salida tendrá una sola fila que resume todas las observaciones de la entrada. Contendrá una columna para cada variable de agrupación y una columna para cada una de las estadísticas de resumen que haya especificado.
## Realizar un conteo de registros por especie
Mammillaria_15 <- Mammillaria_gbif %>%
select(family, species, decimalLatitude, decimalLongitude,
country, stateProvince, year, month, day, verbatimLocality,
recordedBy, identifiedBy) %>%
filter(!is.na(species)) %>%
group_by(species) %>%
count() %>%
arrange(n)
## Obtener el maximo y el minimo valor para la Longiud por estado
Mammillaria_16 <- Mammillaria_gbif %>%
select(family, species, decimalLatitude, decimalLongitude,
country, stateProvince, year, month, day, verbatimLocality,
recordedBy, identifiedBy) %>%
filter(!is.na(stateProvince)) %>%
group_by(stateProvince) %>%
summarise(minlong = min(decimalLongitude), maxlong = max(decimalLongitude))
Ejercicios
Para realizar los ejercicios descargar los siguentes archivos: Altura de tallos, Diametro de tallos, Distancia entre costillas, Ejercicio dplyr.